Octave に有望なコード群を見つけたので、そっちを優先してやることにしました。 Peak Fit With Octave 目標は同じか、それ以上。Python系の情報も一応収集中 Peak Fit With Python また、Gnuplotでさえ、結構できる みたいです。
目標 (Rど素人で、統計も数学も、もう知らんのでヨチヨチ歩きから始める)
ベクモニ、テレミノ、SPE形式のスペクトルを与えると、
- 形式を推定してデータを読む (SPE形式は、既に一応読める。)
- 適当にスムージングして、K40のピークを推測する (一応スムージングと、ピークの検出は、始めた)
- 推測の結果を確認し(あるいは、候補の中から人間に選択させ)、K40のフィットと補正の推測をする。
- 以上の結果から、Cs134,Cs137,Tl208などのフィット。
もしも上の目標が出来たら
- Ra226,Th232の娘の推測とフィット。そして、その情報をK40のフィットや609KeVの推測などに使う。
- FWHMの推測とその確認、利用。
- 核種間の割合の推測と確認。情報の年月日から、Cs134,Cs137の割合を計算して確かめる。
- 校正用線源Eu152などを使って、機器とソフトなどの癖の把握とその利用。
- 分かっている機器などの情報の解析と利用。種類、シンチレーターの大きさ、ケースの材質や厚さ、などなど。
- 機器の特性が分かった状態で、不明核種の判別とフィット。
- あるかもしれない、指定された核種の推測ピークパターンの表示
- テレミノとかのプラグインにする。
- R のWEB実行環境を整える。Mwikiに、Rプラグインをつけると、WEB上でコードをコピペして、自動的にグラフが貼り付けられる、とかすると楽。
- みかげさんのSpviewerに、ピークフィット機能を追加する。(JavaScript実装?R呼び出し?)
やりたいことと、その理由については、新たにまとめページを作りました。 Theremino Mca To Do Ja
やったこと
- RとR Studioをダウンロードしてインストール http://oku.edu.mie-u.ac.jp/~okumura/stat/R-win.html
- いくつかの初歩の学習。 http://oku.edu.mie-u.ac.jp/~okumura/stat/first.html
- findPeaks の為の、 quantmodパッケージと依存してるパッケージのインストール http://cran.r-project.org/web/packages/quantmod/index.html
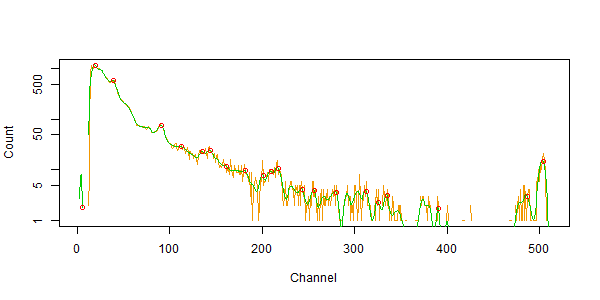
- 奥村さんコードのコピペと組み合わせて、みかげさんの牛乳データでもってスムージングのテスト。
data = read.csv("http://www.mikage.to/radiation/spviewer/data_fnf401/fnf401_milk.csv" , skip=22, nrow=1022, header=FALSE)
names(data) = c("ch","sp")
plot(data$ch, data$sp, type="l", log="y", xlab="Channel", ylab="Count", col="#f39800")
- lowess は、あんまりこれには向いていないようなので、他のものを試す。グラフをLog表示にして、Splineにしてみたら、大分マシ。
# sl=lowess(data$sp, f=0.03, delta=0.5)
require(graphics)
sl=Smooth.spline(data$sp)
lines(sl, col = 3)
library(Defaults)
library(quantmod)
p=findPeaks(sl$y, 0.08)
points(sl$x[p], sl$y[p], col=2)
- とりあえず、プロジェクトを作って、ここまでのコードをファイルに書いて、簡単に実行できるようにして色々試しているところ。
- 「(Log表示の時に、)右からみて、最初のでかい山」って、数学的か統計的か幾何的に、どうRで書けばいいんだろう?
- findpeaks か、類似の奴が、ピークの大きさも持っていれば、それで少しは検討がつく。
- 今は、たまたま最後のピークがK40になっているが、他のデータで色々試さないとわからん。
- となると、SPEファイル形式、テレミノ形式は、自分のデータや、Web上のデータがあるし、これらをまず読み込めるようにしよう。
- それから、ベクモニ形式なんかも読めるようにしよう。ということで、スムージングとピーク検出はちょっと後回しにして、データの自動読み取りをやってみよう。
- まずは、SPEファイル。
fn = "../hogehoge.spe" # 後で、ファイル名で判断して(あるいはファイルを読んでから?)複数形式を読み込む関数か何かにするでの、ファイル名は変数にしておく。
ds = scan(fn, what="") # ファイルを、全部文字としてひとまず読み込む。
i = match("$DATA:",ds)+2 # データの始まりを探す
n = as.integer(ds[i])+1 # データの終わりのインデックスを読む
ne = n + i # いまいち計算が「添え字?」で使えるのか良く分からんので念のため先に計算しておく。
i = i+1 # インクリメントって i++ とかできんのかな?
dsp = as.integer(ds[i:ne]) # データ部分だけ切り出して、整数列に変換。 yがこれ
dch = 1:n # xがこれ。
- 一々やることなすこと検索して探さないとわからんので時間がかかる。次はZIPです。Pythonプログラマーの為のR入門って、どこかに落ちていないのかな?データ構造がいまいちわからん。
- こんなのを読み始めてしまった。とっても元気の出るタイトルだ。 http://lib.stat.cmu.edu/S/Spoetry/Tutor/R_inferno.pdf
- 面白いけど、こんなの読んで本当に分かろうとしたら、いつまで経っても終わらん。通常は、アホで怠け者の初心者が使うアンチョコがどこかに落ちているはずなんだが、Rにはないのか?Ocamlとちょっと雰囲気が似てるぞ。Ocamlは分かり始めると、その後は、なんとかかんとかなったけど。
- そうだ、ロゼッタコードで勉強しよう!これでPythonとか他のコードと比べて睨んでいると、普通は、段々分かってくる。 http://rosettacode.org/wiki/Category:R
- なんだ、ただ data.frame(c1,c2)とやれば良いだけだった。
data = data.frame(dch, dsp) # この後は、最初のcsvファイルを読んだ後と同じ。 names(data) = c("ch","sp")
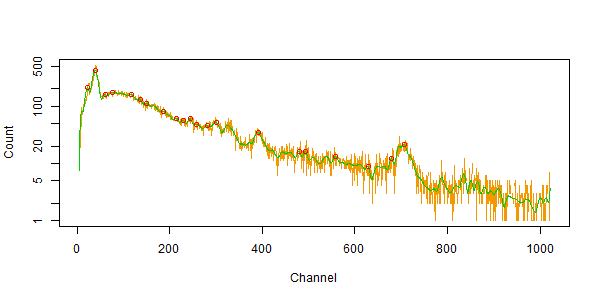
- 早速別のテストデータを読んだら、あんまり芳しくない。Log表示では、ヒストグラムのデータがゼロだとぐちゃぐちゃになる。これでは、ユーザー様に、どのピークをK40として使いましょうか?とお伺いをたてるのも、ちょっと恥ずかしい。大体、人間様が見ても、どれがK40なのか、そんなに明らかではない。多分、225chのが、そうなんだろうと思うが。こうなったら、SPEファイルの補正データを拾ってきて、それを使うのがいいかもしれない。まともなユーザー様なら、機器は、多少は校正されている筈だし。まともでないユーザー様が、どうしようもないスペクトルを食わせたら、「チャンネル番号か、KeVでK40を指定してくださいませ。」と逆に攻めれば良いだけかもしれない。ピーク検出の前に、(分かっている場合、予想できる場合は)FWHMを適度に考慮したスムージングをしないと、初期データのギザギザや、効率の悪い高エネルギー領域などで嘘ピークを検出してしまう。(既知、又は予想される)効率の曲線と、FWHMの対エネルギー曲線も計算に入れないとならないかもしれないけど、もっと、安直で、かつ結果的に、まあまあ同じような結果の出る方法も、あるような気がする。
- さっき、ここで見かけた、正攻法?らしいヒストグラムの曲線化の方法を使ってやって見たほうが良いかも http://ww2.coastal.edu/kingw/statistics/R-tutorials/graphically.html
- まあ、一応ファイルの読み込み方と、Rが使うデータフレームというデータ構造に変換するやり方はわかったし、グラフをプロットして、ファイルに落として、とか、少し分かってきたので、多少気持ちが良い。しかし、こんなの、学生だったら、30分で楽勝でやれるんじゃないか?少しRを学んだ人なら、10分で出来そうなことに、とっても時間がかかってしまった。
- 参考リンク集
http://www.silvertwilight.gr.jp/weblog/archives/2009/10/rdespectrum.html
http://hp.vector.co.jp/authors/VA052342/index.htm
http://ja.wikipedia.org/wiki/%E6%9B%B2%E7%B7%9A%E3%81%82%E3%81%A6%E3%81%AF%E3%82%81
ここは、なかなか充実している。これにコンプトン散乱のパターンを加え、検出器の特性などなどを加味すれば、面白そう。まだ全然分かっていないが、とても素晴らしいのではないか、という気がする。
http://terpconnect.umd.edu/~toh/spectrum/Integration.html
http://terpconnect.umd.edu/~toh/spectrum/TOC.html
http://terpconnect.umd.edu/~toh/spectrum/PeakFindingandMeasurement.htm
http://terpconnect.umd.edu/~toh/spectrum/PeakFindingandMeasurement.htm#ipeak
Mingw版のOctave
Octave は、新しいページでやってみよう。 Peak Fit With Octave
- RはRで使えるものはなんでも使って、色々やってみよう。道具が多いに越したことはない。
これを読んで笑っているあなた、さらさらとRのコードを書いて、メールで私まで送ってください。
nkom あっと pico.dreamhosters.com
2015年現在、やっぱり色々と目移りした後で、Gnuplotで、徐々にやっています。
Peak Fit With Gnuplot これって、色々マシンで動きますし、単純なことしか出来ない様に見えて、
なかなか侮れません。まあ、RやOctave,Pythonなどなどには遠く及びませんが、そういうシステムを
マシンに入れなくても、「Gnuplotだけ」でおおよそ出来てしまいます。
- 後で再利用するかもしれないフィットのコード
fit = gnls(sp ~ c + d*(ch-350) + b*(ch-350)^2
points(data$ch[250:450], fitted(fit), type="l")+ e1*dnorm((ch-m1)/s1)
+ e2*dnorm((ch-m2)/s2)
+ e3*dnorm((ch-m3)/s3),
data=data, subset=250:450,
start=list(c=10,d=-0.05,b=0.001,e1=10,e2=10,e3=10,
m1=300,m2=325,m3=395,s1=7,s2=7,s3=7),
weights=varPower(fixed=0.5),
control=list(nlsTol=1e-4)) # 収束しないときは1e-3に直す
a = coef(fit)
points(data$ch, a['c']+a['d']*(data$ch-350)+a['b']*(data$ch-350)^2, type="l")
奥村さんのサイトよりコピーしてきた使えるかもしれないコード。
http://oku.edu.mie-u.ac.jp/~okumura/stat/peakfit.php
こういうのみんな変数化して、代入する値を変えればコピペで使えるようになっていると
学校時代は数十年前に終わった人間には、楽なんですが。
これらの例だけで大変助かっておりますし、何一つ文句を言えた筋合いでは無いことは承知しておりますが、何分急激に勉強する事が非常に多いので、専門家の方が
モジュール化、関数化しておいていただけると、実装屋としては、ぽこぽこはめ込んで、
組み合わせて、すり合わせて、ごり押しして、なんとかバラバラにならずに動くものを
作りやすいのです。
例題を見て、応用問題を解いて、自分でモジュール化して、というのは、若い人の勉強には
大変素晴らしいことだと思いますが、何分昨今のコピペ文明と歳のせいで、脳みそも
腐っておりますので、ちょっと、と言うかかなり大変です。
library(nlme)
data = read.csv("test95.csv", skip=22, nrow=2040, header=FALSE)
names(data) = c("ch","sp")
plot(data$ch, data$sp, type="l", xlab="Channel", ylab="Count", col="#f39800")
plot(data$ch, data$sp, type="l", xlim=c(1200,1600), ylim=c(0,100),
xlab="Channel", ylab="Count", col="#f39800")
fit = gnls(sp ~ c + d*(ch-1200) + b*(ch-1600)^2
+ e1*dnorm((ch-m1)/s1),
data=data, subset=1200:1600,
start=list(c=10,d=-0.05,b=0.001,e1=10,
m1=1460,s1=7),
weights=varPower(fixed=0.5),
control=list(nlsTol=1e-4)) # 収束しないときは1e-3に直す
points(data$ch[1200:1600], fitted(fit), type="l")
a = coef(fit)
points(data$ch, a['c']+a['d']*(data$ch-1600)+a['b']*(data$ch-1600)^2, type="l")
sum(a['e1']*dnorm((data$ch-a['m1'])/a['s1']))
v = vcov(fit)
sqrt(v['e1','e1']/a['e1']^2 + v['s1','s1']/a['s1']^2 + 2*v['e1','s1']/(a['e1']*a['s1']))
sum((sp[1200:1600]-fitted(fit))^2/fitted(fit))
a = coef(fit2) # 係数 a
sum(a['e']*dnorm((ch-a['m'])/a['s'])) v = vcov(fit2) # 共分散行列 sqrt(v['e','e']/a['e']^2 + v['s','s']/a['s']^2 + 2*v['e','s']/(a['e']*a['s']))
fit = glm(sp ~ ch + dnorm((ch-50)/5), data=data, family=poisson(link="identity"))
summary(fit)
summary(fit)$coefficients[3,1:2]*sum(dnorm((ch-50)/5))
fit = glm(sp ~ ch + dnorm((ch-5000)/500), data=data, family=poisson(link="identity"))
fit = gnls(sp ~ c + d*ch + e*dnorm((ch-5000)/500), data=data, start=list(c=0.5,d=0,e=1), weights=varPower(fixed=0.5))
参考資料置き場
http://oku.edu.mie-u.ac.jp/~okumura/stat/121005.html
https://gist.github.com/koh-t/871963
https://gist.github.com/mickey24/869400